| 铁证如山:石正丽造了Covid-19!(中文翻译) |
| 送交者: 2023年06月02日11:35:59 于 [世界时事论坛] 发送悄悄话 |
|
|
|
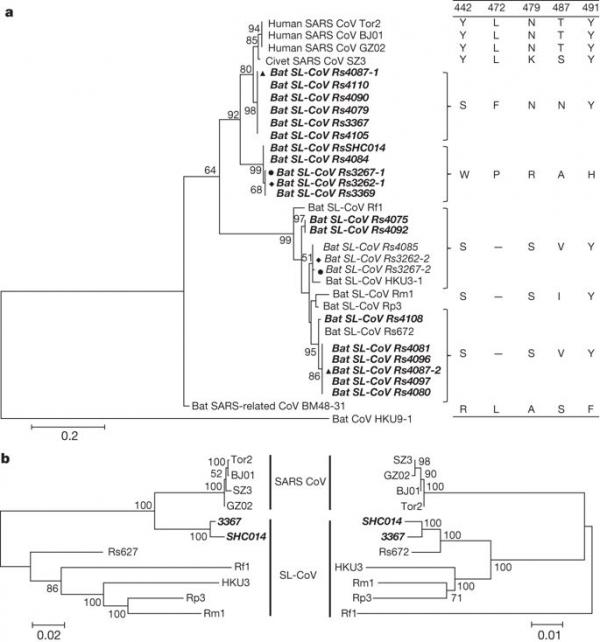
Isolation and characterization of a bat SARS-like coronavirus that uses the ACE2 receptor • Xing-Yi Ge, • Jia-Lu Li, • Xing-Lou Yang, • Aleksei A. Chmura, • Guangjian Zhu, • Jonathan H. Epstein, • Jonna K. Mazet, • Ben Hu, • Wei Zhang, • Cheng Peng, • Yu-Ji Zhang, • Chu-Ming Luo, • Bing Tan, • Ning Wang, • Yan Zhu, • Gary Crameri, • Shu-Yi Zhang, • Lin-Fa Wang, • Peter Daszak & • Zheng-Li Shi(石正丽) Nature 第 503 卷,第 535–538 页(2013)引用这篇文章 • 59k 次访问 • 299 次引用 • 1125 Altmetric • 指标详情 摘要 2002-3 年由严重急性呼吸系统综合症冠状病毒 (SARS-CoV) 引起的大流行是近代历史上最重大的公共卫生事件之一。 中东呼吸综合征冠状病毒 2 的持续爆发表明,这组病毒仍然是一个主要威胁,而且它们的分布范围比以前认识到的要广。 尽管蝙蝠被认为是这两种病毒的天然宿主3、4、5,但从蝙蝠中分离出 SARS-CoV 的祖病毒的尝试并未成功。 现在已经从中国、欧洲和非洲的蝙蝠中报道了多种 SARS 样冠状病毒 (SL-CoV)5,6,7,8,但由于它们与该病毒的系统发育差异以及 他们的刺突蛋白无法使用 SARS-CoV 细胞受体分子,即人血管紧张素转换酶 II (ACE2)9,10。 在这里,我们报告了来自中国云南中国马蹄蝠(家族:Rhinolophidae)的两种新型蝙蝠冠状病毒的全基因组序列:RsSHC014 和 Rs3367。 这些病毒与 SARS-CoV 的关系比之前发现的任何蝙蝠冠状病毒都要密切,特别是在刺突蛋白的受体结合域。 最重要的是,我们首次报告了在 Vero E6 细胞中从蝙蝠粪便样本中分离出活 SL-CoV(蝙蝠 SL-CoV-WIV1)的记录,它具有典型的冠状病毒形态,与 Rs3367 的序列同一性为 99.9%,并使用来自人类的 ACE2, 果子狸和中国马蹄蝙蝠进入细胞。 初步体外试验表明,WIV1 还具有广泛的物种嗜性。 我们的研究结果提供了迄今为止最有力的证据,证明中国马蹄蝠是 SARS-CoV 的天然宿主,中间宿主可能不是某些蝙蝠 SL-CoV 直接感染人类所必需的。 他们还强调了针对新兴疾病热点地区高风险野生动物群体的病原体发现计划作为大流行防范战略的重要性。 主要的 2002-3 年的 SARS1 大流行和中东呼吸综合征冠状病毒 (MERS-CoV)2 的持续出现表明,CoV 是一种重大的公共卫生威胁。 SARS-CoV 被证明使用人类 ACE2 分子作为其进入受体,这被认为是其跨物种传播能力的标志 11。 位于 SARS-CoV 刺突 (S) 蛋白氨基末端区域(氨基酸 318-510)的受体结合域 (RBD) 直接参与与 ACE2 的结合(参考文献 12)。 然而,尽管系统发育证据表明 SARS-CoV 是从蝙蝠 SL-CoV 进化而来的,但所有先前发现的 SL-CoV 在其 S 蛋白的 RBD 中都与 SARS-CoV 存在主要序列差异,包括一两个缺失 6,9。 用 SARS-CoV S 替换一种 SL-CoV S 蛋白的 RBD 赋予了使用人类 ACE2 并在小鼠中有效复制的能力9,13。 然而,迄今为止,还没有从蝙蝠中分离出 SL-CoV,也没有发现源自蝙蝠的野生型 SL-CoV 使用 ACE2。 我们在中国云南省昆明市的一个地点对中华犀牛群落中的 SL-CoV 进行了为期 12 个月的纵向调查(2011 年 4 月至 2012 年 9 月)(扩展数据表 1)。 使用先前公布的方法 5、14 从单个蝙蝠身上收集了总共 117 个肛拭子或粪便样本。 进行了一步逆转录 (RT) 套式 PCR 以扩增 RNA 依赖性 RNA 聚合酶 (RdRP) 基序 A 和 C,它们在甲型冠状病毒和乙型冠状病毒中是保守的15。 117 个样本中有 27 个 (23%) 通过 PCR 被归类为阳性,随后通过测序确认。 如前所述 16,通过细胞色素 b 序列分析,确认所有阳性样品的物种来源为 R. sinicus。 在 10 月收集的样本中观察到更高的患病率(2011 年为 30%,2012 年为 48.7%)高于 4 月(2011 年为 7.1%)或 5 月(2012 年为 7.4%)(扩展数据表 1)。 S 蛋白 RBD 序列分析表明存在七种不同的 SL-CoV 菌株(图 1a 和扩展数据图 1 和图 2)。 除了与先前描述的 SL-CoV(Rs672、Rf1 和 HKU3)5、8、17、18 密切匹配的 RBD 序列外,还发现了两种新菌株(命名为 SL-CoV RsSHC014 和 Rs3367)。 确定了它们的全长基因组序列,发现两者的大小均为 29,787 个碱基对(不包括 poly(A) 尾巴)。 这两个基因组与人类 SARS-CoV(Tor2 株)的整体核苷酸序列同一性为 95%,高于之前在中国(88-92%)5,8,17,18 或欧洲观察到的蝙蝠 SL-CoV( 76%)6(扩展数据表 2 和扩展数据图 3 和图 4)。 在这些新的 SL-CoV 和 SARS-CoV 之间的蛋白质水平上观察到更高的序列同一性(扩展数据表 3 和 4)。 为了解这两种新型 SL-CoV 毒株的进化起源,我们使用重组检测程序 4.0 包 19 使用可用的蝙蝠 SL-CoV 毒株基因组序列(Rf1、Rp3、Rs672、Rm1、HKU3 和 BM48-31)进行了重组分析 以及人类和果子狸的代表性 SARS-CoV 毒株(BJ01、SZ3、Tor2 和 GZ02)。 检测到叁个具有强 P 值 (<10-20) 的断点,并得到相似图和引导扫描分析的支持(扩展数据图 5a、b)。 断点位于 Rs3367(和 RsSHC014)基因组的第 20,827、26,553 和 28,685 位核苷酸,并生成覆盖第 20,827–26,533 位核苷酸(5,727 个核苷酸)的重组片段(包括部分开放阅读框 (ORF) 1b、全长 S、ORF3、 E 和部分 M 基因)和核苷酸 26,534–28,685(2,133 个核苷酸)(包括部分 ORF M、全长 ORF6、ORF7、ORF8 和部分 N 基因)。 使用主要和次要亲本区域的系统发育分析表明,Rs3367 或 RsSHC014 是谱系重组的后代,最终导致 SARS-CoV 和 SL-CoV Rs672(图 1b)。 图 1:基于 S RBD 区域和蝙蝠 SL-CoV Rs3367 或 RsSHC014 的两个亲本区域的氨基酸序列的系统发育树。
a,使用 ClustalW 软件将 SARS-CoV S 蛋白氨基酸残基 310-520 与蝙蝠 SL-CoV 的同源区域对齐。 使用泊松模型构建最大似然系统发育树,其自举值由 MEGA5 软件包中的 1,000 次重复确定。 本研究中确定的 RBD 序列以粗体显示并以样本编号命名。 与人类 ACE2 分子相互作用的关键氨基酸残基显示在树的右侧。 SARS-CoV GZ02、BJ01 和 Tor2 分别从 2003 年 SARS 爆发的早期、中期和晚期患者中分离出来。SARS-CoV SZ3 是在 2003 年从中国广东采集的 Paguma larvata 中鉴定出来的。 SL-CoV Rp3、Rs672 和 HKU3-1 是从中国采集的中华红斑狼蛛中鉴定出来的(分别为:广西,2004 年;贵州,2006 年;香港,2005 年)。 Rf1 和 Rm1 分别从 2004 年在中国湖北采集的 R. ferrumequinum 和 R. macrotis 中鉴定出来。Bat SARS 相关冠状病毒 BM48-31 从 2008 年在保加利亚采集的 R. blasii 中鉴定出来。Bat CoV HKU9-1 是从 2005/2006 年在中国广东收集的 Rousettus leschenaultii 中鉴定出来的,并用作外群。 在当前研究中确定了所有粗体和斜体序列。 实心叁角形、圆形和菱形表示样本被两种不同的 SL-CoV 共同感染。 “-”表示氨基酸缺失。 b,Rs3367 或 RsSHC014 的两个亲本区域的系统发育起源。 最大似然系统发育树由两个片段的比对构建,分别覆盖 Rs3367 基因组的核苷酸 20,827-26,533(5,727 个核苷酸)和 26,534-28,685(2,133 个核苷酸)。 出于显示目的,这些树是中点根的。 分类单元根据菌株名称进行注释:SARS-CoV、SARS 冠状病毒; SARS 样冠状病毒,蝙蝠 SARS 样冠状病毒。 两种新型 SL-CoV,Rs3367 和 RsSHC014,以粗体和斜体显示。 这两种新的 SL-CoV 与之前鉴定的 SL-CoV 之间最显着的序列差异在于其 S 蛋白的 RBD 区域。 首先,它们与 SARS-CoV 具有更高的氨基酸序列同一性(RsSHC014 和 Rs3367 分别为 85% 和 96%)。 其次,没有缺失,它们与 SARS-CoV RBD 区域具有完美的序列比对(扩展数据图 1 和 2)。 结构和诱变研究先前已经确定了 SARS-CoV S 蛋白 RBD 中的五个关键残基(氨基酸 442、472、479、487 和 491),它们在受体结合中具有关键作用 20, 21。 尽管发现 RsSHC014 S 蛋白中的所有五个残基均不同于 SARS-CoV,但 Rs3367 RBD 中的五个残基中有两个是保守的(图 1 和扩展数据图 1)。 尽管在过去十年中蝙蝠 CoV 序列迅速积累,但还没有成功分离病毒的报道 6,22,23。 我们尝试从 SL-CoV PCR 阳性样本中分离出来。 使用优化的方桉和 Vero E6 细胞,我们获得了一个在第二次盲传期间引起细胞病变效应的分离物。 纯化的病毒颗粒在电子显微镜下显示出典型的冠状病毒形态(图 2)。 使用序列非依赖性扩增方法 14 进行序列分析以避免 PCR 引入的污染表明该分离物与 Rs3367 几乎相同,具有 99.9% 的核苷酸基因组序列同一性和 S1 区域的 100% 氨基酸序列同一性。 新分离株被命名为 SL-CoV-WIV1。 图 2:纯化病毒体的电子显微照片。
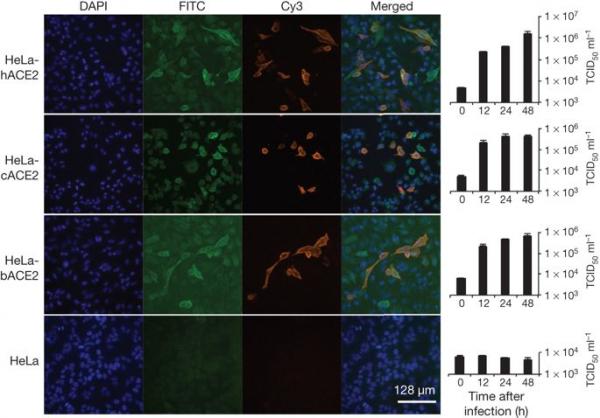
从 10 毫升培养物中收集病毒粒子,固定并通过蔗糖梯度离心法浓缩/纯化。 将沉淀的病毒颗粒悬浮在 100 μl PBS 中,用 2% 磷钨酸 (pH 7.0) 染色,并使用 Tecnai 透射电子显微镜 (FEI) 在 200 kV 下直接检查。 为了确定 WIV1 是否可以使用 ACE2 作为细胞进入受体,我们使用表达或不表达 ACE2 的 HeLa 细胞进行了病毒感染性研究,这些细胞来自人类、果子狸或中国马蹄蝠。 我们发现 WIV1 能够使用不同来源的 ACE2 作为进入受体,并在表达 ACE2 的细胞中有效复制(图 3)。 据我们所知,这是首次鉴定出能够使用 ACE2 作为进入受体的野生型蝙蝠 SL-CoV。 图 3:通过免疫荧光测定和实时 PCR 确定的 SL-CoV-WIV1 受体使用分析。
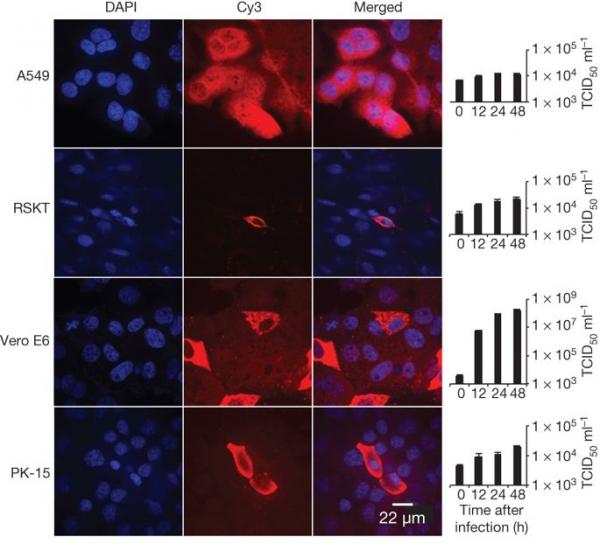
确定表达和不表达 ACE2 的 HeLa 细胞中的病毒感染性。 b、蝙蝠; c、果子狸; 哈,人类。 先用山羊抗人 ACE2 抗体检测 ACE2 表达,然后用异硫氰酸荧光素 (FITC) 偶联的驴抗山羊 IgG 检测。 用针对 SL-CoV Rp3 核衣壳蛋白的兔抗体检测病毒复制,然后用花青 3 (Cy3) 偶联的小鼠抗兔 IgG 检测病毒复制。 细胞核用 DAPI(4',6-二脒基-2-苯基吲哚)染色。 列(从左到右)分别显示细胞核染色(蓝色)、ACE2 表达(绿色)、病毒复制(红色)、合并的叁重染色图像和实时 PCR 结果。 (n = 3); 误差条代表标准偏差。 为了评估其跨物种传播潜力,我们对一系列物种的细胞系进行了感染性测定。 我们的结果(图 4 和扩展数据表 5)表明蝙蝠 SL-CoV-WIV1 可以在人肺泡基底上皮细胞 (A549)、猪肾 15 (PK-15) 和 Rhinolophus sinicus 肾 (RSKT) 细胞系中生长,但是 不存在于人子宫颈 (HeLa)、叙利亚金仓鼠肾 (BHK21)、鼠耳鼠肾 (BK)、中华鼠尾草肾 (MCKT)、Rousettus leschenaulti 肾 (RLK) 或 Pteropus alecto 肾 (PaKi) 细胞系中。 实时 RT-PCR 表明 WIV1 在 A549、PK-15 和 RSKT 细胞中的复制效率远低于在 Vero E6 细胞中的复制效率(图 4)。 图 4:通过免疫荧光测定和实时 PCR 确定的 SL-CoV-WIV1 宿主范围分析。
A549、RSKT、Vero E6 和 PK-15 细胞中的病毒感染。 如图 3 所述检测病毒复制。列(从左到右)分别显示细胞核染色(蓝色)、病毒复制(红色)、合并的双染色图像和实时 PCR 结果。 n = 3; 误差线代表 s.d. 为了评估人 SARS-CoV 血清对 WIV1 的交叉中和活性,我们使用 2003 年收集的 SARS 患者的九份恢复期血清进行了血清中和试验。结果表明,其中七份能够完全中和 100 组织培养感染剂量 50 (TCID50) WIV1 以 1:10 至 1:40 的稀释度,进一步证实了 WIV1 与 SARS-CoV 之间的密切关系。 我们的研究结果对公共卫生具有重要意义。 首先,它们提供了迄今为止最明确的证据表明 SARS-CoV 起源于蝙蝠。 我们之前的工作提供了这方面的系统发育证据 5,但直到现在,由于缺乏蝙蝠 SL-CoV 可以自然感染人类细胞的分离物或证据,人们对这一假设产生了怀疑。 其次,SL-CoV 缺乏使用 ACE2 受体的能力以前被认为是它们直接溢出到人类的主要障碍,这支持了在 SARS 爆发期间果子狸是 SARS-CoV 适应人类传播的中间宿主的观点 24 . 然而,SL-CoV-WIV1 使用人类 ACE2 的能力反驳了 SL-CoV-WIV1 这一步的必要性,并表明蝙蝠直接感染人类是某些蝙蝠 SL-CoV 的合理场景。 面对最近发现的具有广泛地理分布的多样化且不断增长的类 SARS 冠状病毒可能溢出,这对公共卫生控制措施产生了影响。 我们的研究结果表明,蝙蝠冠状病毒的多样性大大高于之前报道的多样性。 在这项研究中,我们能够证明在 12 个月的时间里,至少七种不同的 SL-CoV 菌株在单个 R. sinicus 菌落内循环。 该菌落内 SL-CoV 的高度遗传多样性反映在不同菌株对 ACE2 的差异使用中的高度表型多样性。 因此,如果进一步的监测发现能够使用 ACE2 的蝙蝠 SL-CoV 种类繁多,其中一些与 SARS-CoV 的同源性可能比 SL-CoV-WIV1 更接近,也就不足为奇了。 我们的结果——除了最近在沙特阿拉伯蝙蝠身上发现的 MERS-CoV25,以及在中国、非洲、欧洲和北美 3,26,27 中与 MERS-CoV 密切相关的蝙蝠 CoV——表明蝙蝠冠状病毒仍然是一个重要的 对全球公共卫生的威胁。 最后,这项研究证明了针对野生动物的病原体发现计划的公共卫生重要性,这些计划旨在识别“已知的未知数”——与已知病原体密切相关的以前未知的病毒株。 这些项目专注于特定的高风险野生动物群体和疾病出现的热点地区,可能是未来预测、准备和预防大流行病出现的全球战略的重要组成部分 28。 方法总结 如前所述 14,在病毒运输介质中收集咽喉和粪便拭子或新鲜粪便样本。 所有 PCR 均使用 One-Step RT-PCR 试剂盒 (Invitrogen) 进行。 如前所述,针对 RdRP 基因高度保守区域的引物用于检测所有甲型冠状病毒和乙型冠状病毒 15。 简并引物是根据所有可用的 SARS-CoV 和 SL-CoV 基因组序列设计的,用于扩增 S 基因的 RBD 序列或全长基因组序列。 如前所述,简并引物用于扩增蝙蝠 ACE2 基因 29。 将 PCR 产物凝胶纯化并克隆到 pGEM-T Easy Vector (Promega) 中。 至少对四个独立的克隆进行测序以获得共有序列。 PCR 阳性粪便样本(在 200μl 缓冲液中)在 3,000–12,000g 下梯度离心,上清液在 DMEM 中以 1:10 稀释,然后添加到 Vero E6 细胞中。 在 37°C 孵育 1h 后,取出接种物并用含有 2% FCS 的新鲜 DMEM 代替。 细胞在 37°C 下孵育,并每天检查细胞病变效应。 来自不同来源的细胞系在 24 孔板的盖玻片上生长,并以 10 的感染复数接种新型 SL-CoV。感染后 24 小时,使用针对 SL-CoV Rp3 核衣壳蛋白的兔抗体检测病毒复制 其次是 Cy3 缀合的山羊抗兔 IgG。 在线方法 采样 如前所述,蝙蝠被困在它们的自然栖息地中 5。 咽喉和粪便拭子样本收集在病毒转运培养基 (VTM) 中,该培养基由 Hank 平衡盐溶液组成,pH 7.4,含有 BSA (1%)、两性霉素 (15 μg ml−1)、青霉素 G (100U ml−1) 和 链霉素 (50μgml−1)。 为了收集新鲜的粪便样本,每晚约 18:00 将 2.0 x 2.0 米的干净塑料布放在已知的蝙蝠栖息地下方。 第二天早上大约 05:30–06:00 从床单上收集相对新鲜的粪便样本,并将其放入 VTM 中。 样品被运送到实验室并储存在-80℃直至使用。 所有被困在这项研究中的动物在收集样本后都被释放回它们的栖息地。 所有采样过程均由兽医根据与加州大学戴维斯分校的机构间协议(加州大学戴维斯分校协议编号 16048)获得武汉病毒研究所动物伦理委员会 (WIVH05210201) 和生态健康联盟的批准。 RNA提取、PCR和测序 按照制造商的说明,使用病毒 RNA 迷你试剂盒(Qiagen)从 140μl 拭子或粪便样本中提取 RNA。 RNA 在 60μl 无 RNAse 缓冲液(缓冲液 AVE,Qiagen)中洗脱,然后等分并储存在 -80℃。 一步 RT-PCR(Invitrogen)用于检测冠状病毒序列,如前所述 15。 第一轮 PCR 在含有 12.5μl PCR 2× 反应溷合物缓冲液、10pmol 每种引物、2.5mM MgSO4、20U RNase 抑制剂、1μl SuperScript III/Platinum Taq 酶溷合物和 5μl RNA 的 25μl 反应溷合物中进行第一轮 PCR . RdRP 基因片段的扩增如下进行:50°C 30min,94°C 2min,然后进行 40 个循环,包括 94°C 15s,62°C 15s,68°C for 40s,最后 68°C 延伸 5min。 第二轮 PCR 在含有 2.5μl PCR 反应缓冲液、5pmol 每种引物、50mM MgCl2、0.5mM dNTP、0.1μl Platinum Taq 酶(Invitrogen)和 1μl 第一轮 PCR 产物的 25μl 反应溷合物中进行。 RdRP 基因片段的扩增如下进行:94°C 5min,然后进行 35 个循环,包括 94°C 30s,52°C 30s,72°C 40s,最后延伸 72°C 5min。 为了扩增 RBD 区域,使用基于可用的 SARS-CoV 或蝙蝠 SL-CoV 设计的引物进行一步 RT-PCR(第一轮 PCR 引物;F,正向;R,反向:CoVS931F-5'-VWGADGTTGTKAGRTTYCCT-3 '和CoVS1909R-5'-TAARACAVCCWGCYTGWGT-3';第二个PCR引物:CoVS951F-5'-TGTKAGRTTYCCTAAYATTAC-3'和CoVS1805R-5'-ACATCYTGATANARAACAGC-3')。 第一轮 PCR 在 25 μl 反应溷合物中进行,如上所述,除了使用对 S 基因特异的引物。 S 基因 RBD 区域的扩增如下进行:50°C 30min,94°C 2min,然后 94°C 15s,43°C 15s,68 个循环 35 个循环 °C 90s,最后延伸 68°C 5min。 第二轮 PCR 在含有 2.5μl PCR 反应缓冲液、5pmol 每种引物、50mM MgCl2、0.5mM dNTP、0.1μl Platinum Taq 酶(Invitrogen)和 1μl 第一轮 PCR 产物的 25μl 反应溷合物中进行。 扩增如下进行:94°C 5min,然后 40 个循环,包括 94°C 30s,41°C 30s,72°C 60s,最后延伸 72°C 5min 将 PCR 产物凝胶纯化并克隆到 pGEM-T Easy Vector (Promega) 中。 对至少四个独立的克隆进行测序以获得每个扩增区域的共有序列。 全长基因组测序 简并冠状病毒引物是根据GenBank中所有可用的SARS-CoV和蝙蝠SL-CoV序列设计的,特异性引物是根据本研究前几轮测序产生的基因组序列设计的(引物序列将根据要求提供)。 所有 PCR 均使用 One-Step RT-PCR 试剂盒 (Invitrogen) 进行。 分别使用 5' 或 3' RACE 试剂盒 (Roche) 确定 5' 和 3' 基因组末端。 将 PCR 产物凝胶纯化并直接测序或在克隆到 pGEM-T Easy Vector (Promega) 后测序。 至少对四个独立的克隆进行测序,以获得每个扩增区域的共有序列,并且每个区域至少测序两次。 序列分析和数据库登录号 使用 DNAStar 或 Geneious 进行常规序列管理和分析。 使用 ClustalW、BioEdit 或 GeneDoc 进行序列比对和编辑。 基于蛋白质序列的最大似然系统发育树是使用泊松模型构建的,其引导值由 MEGA5 软件包中的 1,000 次重复确定。 本研究中获得的序列已保藏在 GenBank 中,登记号在括号中: WIV1 S (KC881007) 的全长序列; RBD(KC880984-KC881003); ACE2 (KC8810040)。 本研究中使用的 SARS-CoV 序列:人类 SARS-CoV 毒株 Tor2 (AY274119)、BJ01 (AY278488)、GZ02 (AY390556) 和果子狸 SARS-CoV 毒株 SZ3 (AY304486)。 本研究中使用的蝙蝠冠状病毒序列:Rs672 (FJ588686)、Rp3 (DQ071615)、Rf1 (DQ412042)、Rm1 (DQ412043)、HKU3-1 (DQ022305)、BM48-31 (NC_014470)、HKU9-1 (NC_009021)、HKU4 (NC_009019)、HKU5 (NC_009020)、HKU8 (DQ249228)、HKU2 (EF203067)、BtCoV512 (NC_009657)、1A (NC_010437)。 本研究中使用的其他冠状病毒序列:HCoV-229E (AF304460)、HCoV-OC43 (AY391777)、HCoV-NL63 (AY567487)、HKU1 (NC_006577)、EMC (JX869059)、FIPV (NC_002306)、PRCV (DQ811787)、BWCoV (NC_010646)、MHV (AY700211)、IBV (AY851295)。 蝙蝠ACE2基因的扩增、克隆与表达 在 pcDNA3.1 中构建人和果子狸 ACE2 的表达克隆之前已有描述29。 Bat ACE2 是从 R. sinicus(样品编号 3357)中扩增出来的。 简而言之,使用 RNeasy Mini Kit (Qiagen) 从蝙蝠直肠组织中提取总 RNA。 通过用随机六聚体逆转录从总 RNA 合成第一链互补 DNA。 使用正向引物 bAF2 和反向引物 bAR2 扩增全长蝙蝠 ACE2 片段(参考文献 29)。 用KpnI和XhoI将ACE2基因克隆到pCDNA3.1中,测序验证。 将纯化的 ACE2 质粒转染至 HeLa 细胞。 24 小时后,表达人、果子狸或蝙蝠 ACE2 的 HeLa 细胞裂解物通过蛋白质印迹或免疫荧光测定法进行确认。 蛋白质印迹分析 细胞裂解物或含有假病毒的过滤上清液通过 SDS-PAGE 分离,然后转移到硝酸纤维素膜 (Millipore)。 为了检测 S 蛋白,将膜与兔抗 Rp3 S 片段(氨基酸 561–666)多抗体(1:200)一起孵育,并通过碱性磷酸酶 (AP) 偶联的山羊抗兔 IgG 检测结合的抗体 (1:1,000)。 为了检测上清液中的 HIV-1 p24,使用抗 HIV p24 单克隆抗体 (p24 MAb) 作为一抗,稀释度为 1:1,000,然后与 AP 偶联山羊抗小鼠 IgG 以相同稀释度孵育。 为了检测 HeLa 细胞中 ACE2 的表达,使用抗人 ACE2 胞外域的山羊抗体(1:500)作为一抗,然后与辣根过氧化物酶偶联的驴抗山羊 IgG(1:1,000)孵育。 病毒分离 Vero E6 细胞单层保存在补充有 10% FCS 的 DMEM 中。 PCR 阳性样品(在 200μl 缓冲液中)以 3,000–12,000g 梯度离心,上清液在 DMEM 中按 1:10 稀释,然后加入 Vero E6 细胞。 在 37°C 孵育 1h 后,取出接种物并用含有 2% FCS 的新鲜 DMEM 代替。 将细胞在 37°C 下孵育 3 天,并每天检查细胞病变效应。 双剂量叁联抗生素青霉素/链霉素/两性霉素 (Gibco) 包含在所有组织培养基中(青霉素 200 IU ml−1,链霉素 0.2 mg ml−1,两性霉素 0.5 μg ml−1)。 每个样品进行叁个盲传代。 每次传代后,使用靶向 RdRP 或 S 基因的引物,通过 RT-PCR 检查培养物上清液和细胞沉淀中是否存在病毒。 收集上清液 (10ml) 中的病毒粒子并使用 0.1% 甲醛固定 4h,然后使用 Ty90 转子 (Beckman) 通过 20% 蔗糖垫 (5ml) 以 80,000g 超速离心 90min 进行浓缩。 将沉淀的病毒颗粒悬浮在 100 μl PBS 中,用 2% 磷钨酸 (pH 7.0) 染色,并使用 Tecnai 透射电子显微镜 (FEI) 在 200 kV 下进行检查。 免疫荧光法检测病毒传染性 扩展数据表 5 总结了用于本研究的细胞系及其培养条件。通过细胞病变效应 (CPE) 计数确定 Vero E6 细胞中的病毒滴度。 来自不同来源的细胞系和表达来自人类、果子狸或中国马蹄蝠的 ACE2 的 HeLa 细胞在 24 孔板(康宁)的盖玻片上生长,与蝙蝠 SL-CoV-WIV1 一起孵育,感染复数 = 10,持续 1 小时。 取出接种物并用 PBS 洗涤两次并补充培养基。 无 ACE2 表达的 HeLa 细胞和 Vero E6 细胞分别用作阴性和阳性对照。 感染后 24 小时,用 PBS 洗涤细胞,并在 4°C 下用含 4% 甲醛的 PBS (pH 7.4) 固定 20 分钟。 使用山羊抗人 ACE2 免疫球蛋白 (R&D Systems) 随后使用 FITC 标记的驴抗山羊免疫球蛋白 (PTGLab) 检测 ACE2 表达。 使用针对 SL-CoV Rp3 核衣壳蛋白的兔抗体,然后使用 Cy3 缀合的小鼠抗兔 IgG 检测病毒复制。 细胞核用 DAPI 染色。 使用 FV1200 共聚焦显微镜 (Olympus) 检查染色模式。 通过实时 RT-PCR 检测病毒感染性 表达或不表达不同来源的 ACE2 的 Vero E6、A549、PK15、RSKT 和 HeLa 细胞接种 0.1 TCID50 WIV-1,并在 37°C 下孵育 1h。 除去接种物后,将细胞用含有1%FBS的培养基培养。 在 0、12、24 和 48h 收集上清液。 按照制造商的说明,使用病毒 RNA 迷你试剂盒(Qiagen)从 140μl 每种上清液中提取 RNA,并在 60μl 缓冲液 AVE(Qiagen)中洗脱。 在 ABI StepOne 系统上使用 TaqMan AgPath-ID 一步式 RT-PCR 试剂盒(Applied Biosystems)在 25μl 反应溷合物中对 RNA 进行定量,其中含有 4μl RNA,1 × RT-PCR 酶溷合物,1⟩×⟩RT-PCR 缓冲液、40pmol 正向引物 (5'-GTGGTGGTGACGGCAAAATG-3')、40pmol 反向引物 (5'-AAGTGAAGCTTCTGGGCCAG-3') 和 12pmol 探针 (5'-FAM-AAAGAGCTCAGCCCCAGATG-BHQ1-3')。 扩增参数为 50°C 10min、95°C 10min 以及 95°C 15s 和 60°C 20s 的 50 个循环。 来自纯化的 WIV-1 原液的 RNA 稀释液用作标准。 血清中和试验 SARS 患者血清在 56°C 下灭活 30min,然后用于病毒中和测试。 血清从 1:10 开始稀释,然后在 96 孔细胞板中连续两倍稀释至 1:40。 将每 100μl 血清稀释液与 100μl 含有 100μl WIV1 TCID50 的病毒上清液溷合,并在 37℃ 下孵育 1h。 将溷合物一式叁份添加到铺有单层 Vero E6 细胞的 96 孔细胞板的孔中,并在 37°C 下进一步孵育 2 天。 来自健康献血者的血清在每个实验中用作阴性对照。 接种后 2 天使用倒置显微镜观察 CPE。 中和抗体滴度被读取为完全抑制感染孔中 CPE 的血清的最高稀释度。 中和试验重复两次。 重组分析 使用 Clustal X 将 SL-CoV Rs3367 或 RsSHC014 的全长基因组序列与选定的 SARS-CoV 和蝙蝠 SL-CoV 的基因组序列进行比对。使用重组检测程序 (RDP) 4.0 初步扫描比对序列的重组事件(参考文献 1)。 19). 由于 RDP 具有强 P 值 (<10–20),因此通过 Simplot 3.5.1 中实施的相似图和引导扫描分析进一步研究了 RDP 提示的潜在重组事件。 Rs3367 或 RsSHC014 的主要和次要亲本区域的系统发育起源是根据所选 SARS-CoV 和 SL-CoV 的主要和次要亲本区域的基本 ORF 的串联序列构建的。 使用 ClustalX 独立比对了叁个估计断点(20,827–26,553 和 26,554–28,685)之间的两个基因组区域,并生成了 5,727 个碱基对和 2,133 个碱基对的两个比对。 这两个比对用于构建最大似然树以更好地推断片段父代。 本研究中的所有核苷酸编号均基于 Rs3367 基因组位置。 注册码 种质 基因库/EMBL/DDBJ • KC880984 • KC881003 • KC881004 • KC881005 • KC881007 数据存款 叁种蝙蝠 SL-CoV 基因组、蝙蝠 SL-CoV RBD 和 R. sinicus ACE2 基因的序列已保存在 GenBank 中,登录号为 KC881005–KC881007(分别来自 SL-CoV RsSHC014、Rs3367 和 W1V1 的基因组)、KC880984–KC881003( bat SL-CoV RBD 基因)和 KC881004(R. sinicus ACE2)。 |
|
|
 |
 |
| 实用资讯 | |
|
|
| 一周点击热帖 | 更多>> |
| 一周回复热帖 |
| 历史上的今天:回复热帖 |
| 2022: | 郭文贵假仁假义 | |
| 2022: | 郭文贵被围剿 | |
| 2021: | 再谈澳籍华人杨恒均受审 | |
| 2021: | 不要闭门造车搞纯宗教——与张国堂商榷 | |
| 2020: | 巴托勒枚与1550年的巴利亚多利德辩论(1 | |
| 2020: | 安提法ANTIFA到底是个什么组织? | |
| 2019: | 团结起来,争取更大的胜利 | |
| 2019: | 帝国主义、修正主义和一切反动派都是纸 | |
| 2018: | 北京街上,满眼都是黑衣人 | |
| 2018: | 跟着美军上战场:真打起来,我们很可能 | |